上海高研院在mRNA药物递送系统脂质纳米颗粒(LNPs)人工智能可解释性预测模型方面取得重要进展
mRNA药物在疫苗、癌症免疫疗法等领域具有重要的研究价值,mRNA-LNPs的筛选是一项非常有挑战的任务。人工智能赋能科学研究(AI for Science)技术的兴起给mRNA-LNPs快速筛选带来新手段,利用人工智能模型构建分子三维微观结构与生化性质间的映射关系,实现生化性质的精准预测,是未来快速设计自主高转染效率的mRNA-LNPs的重要途径。
针对上述问题,上海高等研究院工业人工智能刘立庄研究团队在mRNA药物递送系统脂质纳米颗粒人工智能可解释性预测模型方面取得重要进展,提出了一种基于Transformer和Mamba的多模态可解释的性质预测模型(TransMA),结果是当前最优,相关研究成果以“TransMA: an explainable multi-modal deep learning model for predicting properties of ionizable lipid nanoparticles in mRNA delivery”为题发表在生物信息期刊Briefings In Bioinformatics上。
本研究设计基于Transformer和Mamba的多模态可解释的性质预测模型,称为TransMA(图1)。TransMA针对分子三维和一维信息多模态特征提取和交互,设计Transformer和状态空间Mamba融合架构;结合均方误差(MSE)损失函数和三元组损失函数,提出一种自适应混合损失函数,对于多模态特征融合提升预测精度具有重要价值;在特征对齐和融合方面,提出一种分子通道注意力机制,建模出分子特征通道之间的相互依赖性,评估原子级注意力分数,识别关键分子结构。TransMA识别出的高注意力分数的原子与实际影响性质的关键原子是一致的(图2),实现模型可解释性,同时,TransMA实现预测均方误差MSE为3.64,决定系数(𝑅²)0.49,对比多种基于图卷积神经网络和Transformer架构,结果显示(图3),在均方误差、决定系数R2和皮尔逊相关系数评价指标中性能最优,指标再次达到该领域最优水平。
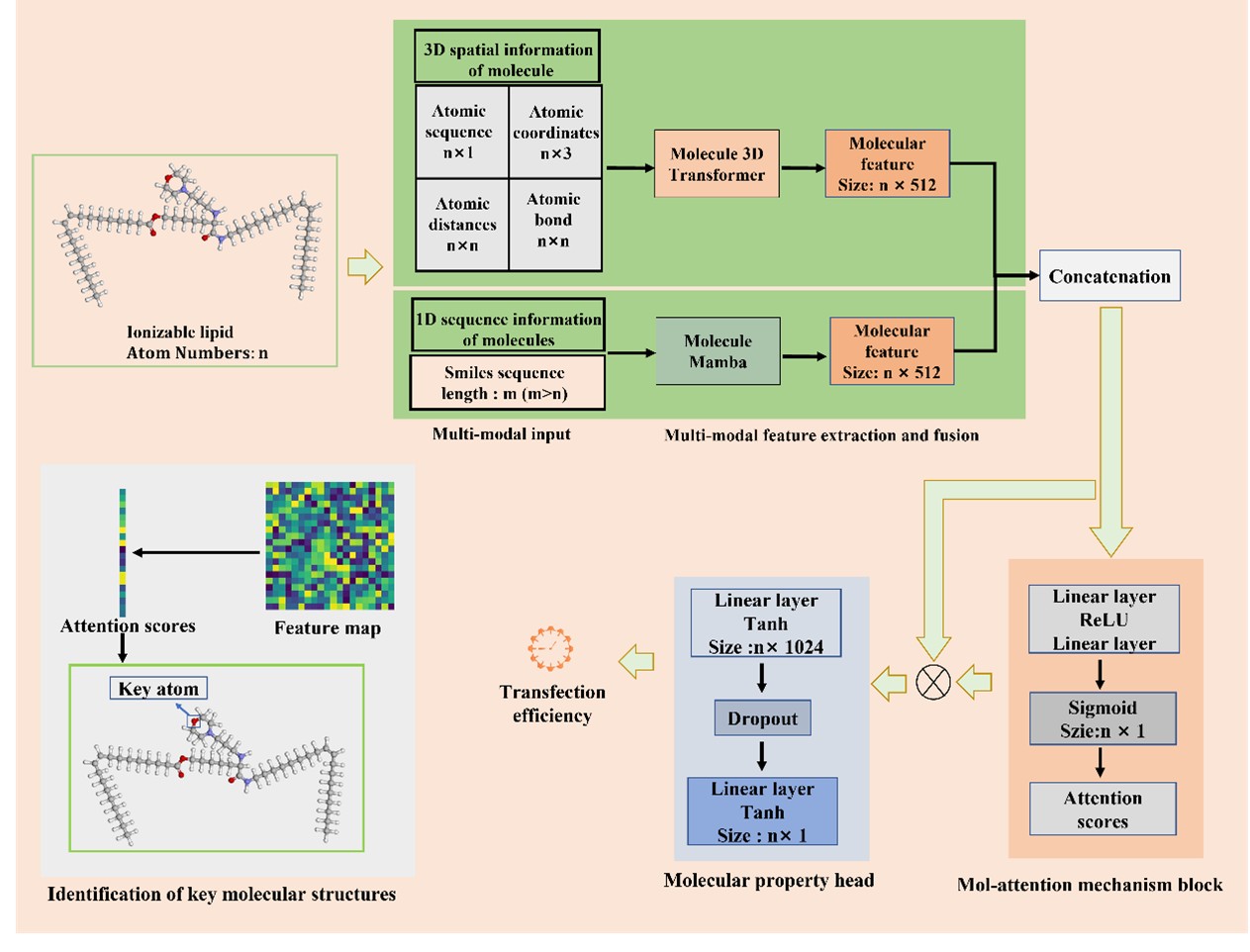
图1 TransMA架构图
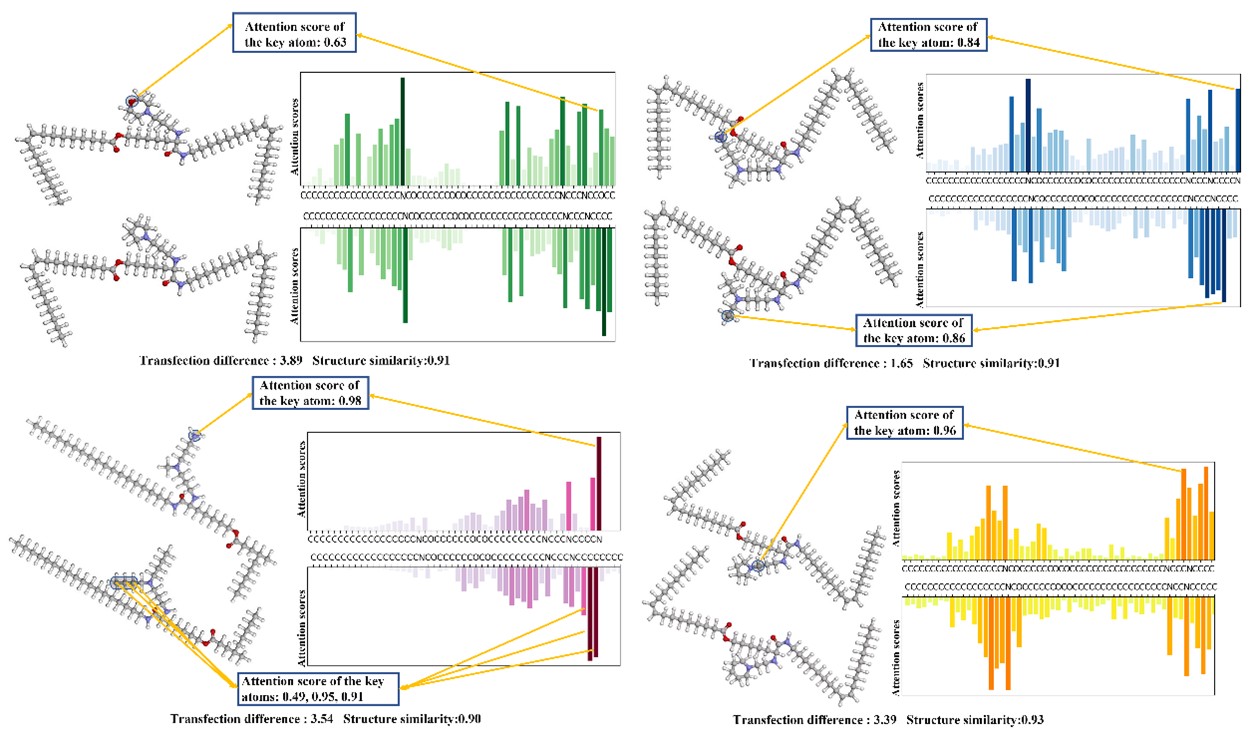
图2 TransMA识别出的高注意力分数的原子与实际影响性质的关键原子对应图
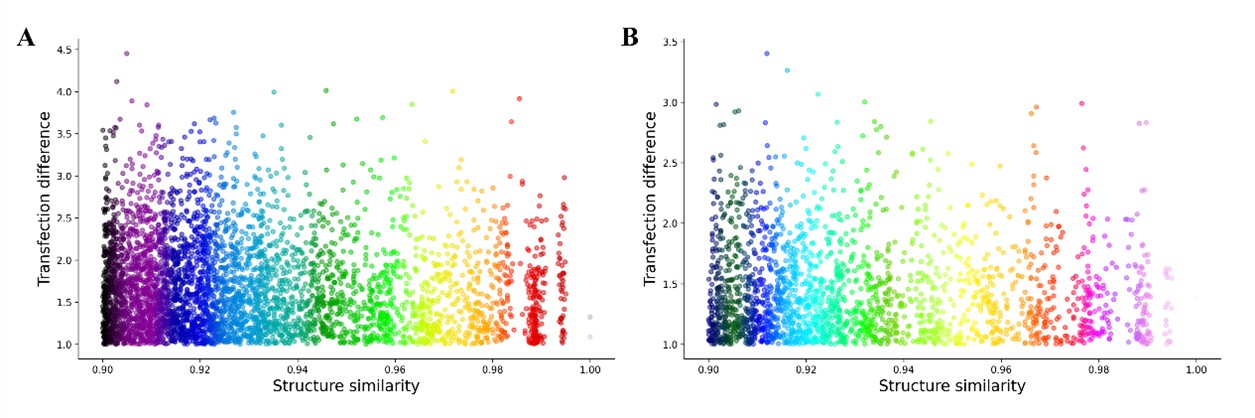
图3 TransMA与多种基于图卷积神经网络和Transformer架构结果对比图
该工作实现快速精准预测mRNA-LNPs的转染效率,能够快速筛选出稳定有效的LNPs,提高mRNA药物递送效率,为多种疾病的个性化治疗提供可靠研究方法和工具,新方法将推动mRNA药物在基因治疗、疫苗开发和药物递送等领域的应用发展。论文的第一作者是高研院硕士研究生武坤、通讯作者刘立庄正高级工程师,联合了日本筑波大学王梓旭博士、华中师范大学杨秀隆博士和意大利热那亚大学Fulvio Mastrogiovanni教授共同完成。
文章链接:https://doi.org/10.1093/bib/bbaf307
附件下载: